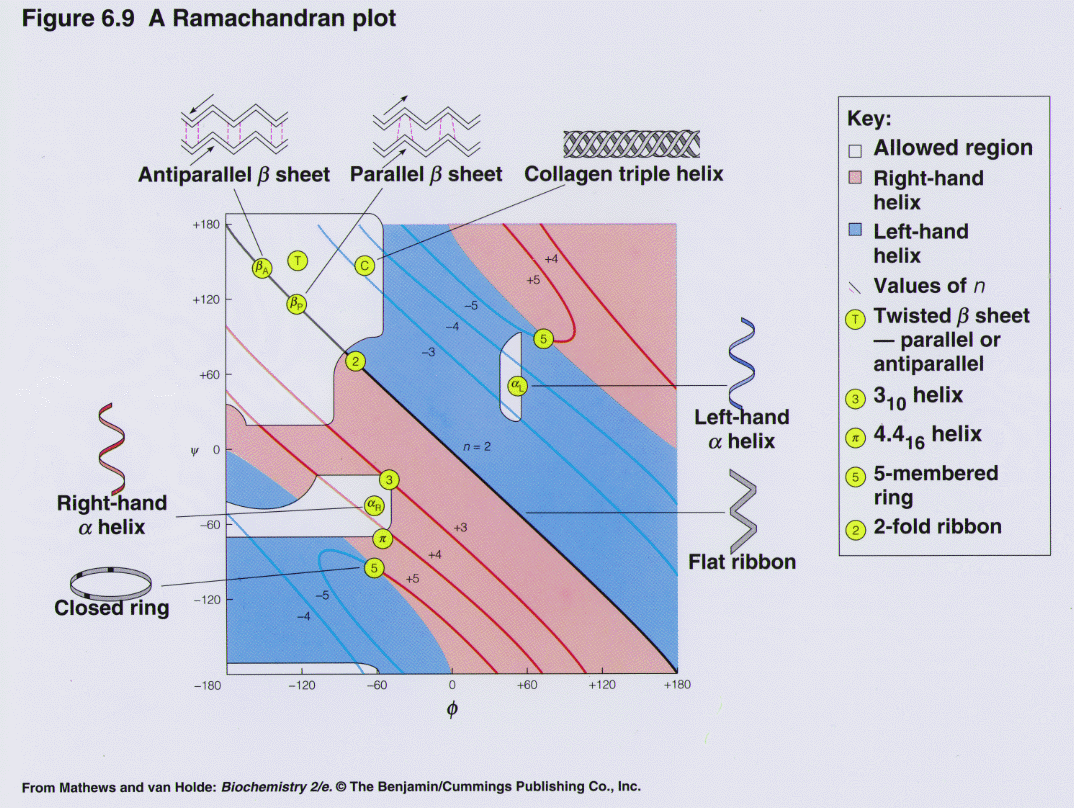
ラマチャンドランプロット (Ramachandran plot) は、1963年にG・N・ラマチャンドラン、C. Ramakrishnan、V. Sasisekharanによって開発された、タンパク質構造中のアミノ酸残基の主鎖二面角φに対してエネルギー的に許容されるψの領域を可視化する手法である。ラマチャンドランダイアグラム (Ramachandran diagram)、[φ,ψ]プロットとしても知られる。左の図では、主鎖二面角φとψのの定義が示されている(ラマチャンドランはφとφ' と呼んでいた)。部分的二重結合性がペプチドを平面に保つため、ペプチド結合のω角は通常180° である。右上の図は、ラマチャンドランらによる1963年と1968年の剛体球計算に基づく、許容されるφとψの主鎖の立体配座の領域を示している。実線で囲まれた領域は標準的な剛体球半径の条件、破線は半径をより小さくした条件、点線はτ (N-Cα-C) 角を緩和した条件で許容される領域が示されている。二面角の値の0° と360° は同一なので、ラマチャンドランプロットの左右と上下の端は連続的である。例えば、プロットの左下に存在する小さな帯状の領域は左上の大きな領域から連続しているものである。
利用
ラマチャンドランプロットは、いくぶん異なる2つの方法で用いられる。1つ目は、右上の図ように、タンパク質中のアミノ酸残基が理論上とりうるψとφ角の値、または立体配座を示すことである。2つ目は、右の図のように構造の検証のために1つの構造中でみられる実験的データポイントの分布を示したり、または下の図のように多数の構造のデータベース中での分布を示したりすることである。
アミノ酸による傾向
より大きな側鎖にはより多くの制限があり、そのためラマチャンドランプロットで許容される領域はより小さくなると予想されるかもしれないが、側鎖による影響は小さい。実際には、Cβにメチレン基が存在するかどうかが大きな影響を与えている。グリシンの側鎖は水素原子だけであり、そのファンデルワールス半径は他の全てのアミノ酸のCH3、CH2やCH基よりもずっと小さく、したがって制限は最も少ない。このことはグリシンについてのラマチャンドランプロットからも明らかであり、許容される領域はかなり広い (下のギャラリーを参照)。対照的に、プロリンは五員環からなる側鎖がCαと主鎖のNを連結しており、そのラマチャンドランプロットは可能なψとφの値の組み合わせが限られていることを示している。プロリンの前に位置する残基も、一般的な場合と比べて値は限定される。
近年の更新
最初のラマチャンドランプロットは、その結論は短いペプチドの結晶学に基づいていたものの、最初の原子分解能のタンパク質構造(1960年に解かれたミオグロビンの構造)が決定されたすぐ後に計算された。それから何十年も経過した現在では、X線結晶構造解析によって決定された数万の高分解能タンパク質構造が存在し、蛋白質構造データバンク (PDB) に登録されている。このデータを利用して、多くの研究でより詳細で正確なφとψのプロットが作成されている。
下の4つの図では、高分解能構造の巨大なデータセットから得られた φ と ψ のデータポイントと、好まれる立体配座の領域と許容される領域の輪郭が、一般的な残基(グリシン、プロリン、プロリンの前を除くすべてのアミノ酸)、グリシン、プロリン、プロリンの前の残基に分けて示されている。図中のラベル、αはα-ヘリックス、Lα は左巻きヘリックス、βはβ-シート、ppIIはポリプロリンIIヘリックスの一般的な位置を表している。このようなクラスタリングはABEGOシステムによって記述されることもある。ABEGOの各文字は、α-ヘリックスと310-ヘリックス、右巻きβ-シートと伸展構造 (extended structure)、左巻きヘリックス、左巻きシート、プロリンで稀にみられるプロットできないシスペプチドを表している。このシステムはモチーフの分類に利用され、近年ではタンパク質の設計にも利用されている。
教科書でペプチド結合の構造的な性質を説明するためにラマチャンドランプロットが利用されることがある一方で、ラマチャンドランプロットの各位置においてペプチドがどのように振る舞うかについての詳細な研究は、近年になって発表されたばかりである。
インド理科大学院のMolecular Biophysics Unitはラマチャンドランプロットの50周年を記念して、International Conference on Biomolecular Forms and Functionsを2013年1月8–11日に開催した。
関連手法
多糖や他の多量体の二面角についても同様のプロットを作成することができる (CARPなど)。タンパク質の側鎖間の二面角に関する同様のプロットにJanin Plotがある。
ギャラリー
データはから。
ソフトウェア
- Web-based Structural Analysis tool for any uploaded PDB file, producing Ramachandran plots, computing dihedral angles and extracting sequence from PDB
- Web-based tool showing Ramachandran plot of any PDB entry
- MolProbity web service that produces Ramachandran plots and other validation of any PDB-format file
- SAVES (Structure Analysis and Verification) — uses WHATCHECK, PROCHECK, and does its own internal Ramachandran Plot
- STING
- PyMOL with the DynoPlot extension
- VMD, distributed with dynamic Ramachandran plot plugin
- WHAT CHECK, the stand-alone validation routines from the WHAT IF software
- UCSF Chimera, found under the Model Panel.
- Sirius
- Swiss PDB Viewer
- TALOS
- Zeus molecular viewer — found under "Tools" menu, high quality plots with regional contours
- Procheck
- Neighbor-Dependent and Neighbor-Independent Ramachandran Probability Distributions
脚注
関連文献
- Richardson, J.S. (1981). “Anatomy and Taxonomy of Protein Structures”. Advances in Protein Chemistry. Advances in Protein Chemistry 34: 167–339. doi:10.1016/S0065-3233(08)60520-3. ISBN 9780120342341. PMID 7020376. , available on-line at Anatax
- Branden, C.-I.; Tooze, J. (1991), Introduction to Protein Structure, Garland Publishing, NY, ISBN 0-8153-0344-0
外部リンク
- DynoPlot in PyMOL wiki
- Link to Ramachandran Plot Map of alpha-helix and beta-sheet locations
- Link to Ramachandran plot calculated from protein structures determined by X-ray crystallography compared to the original Ramachan.
- Proteopedia Ramachandran Plot